

Clontech是cDNA合成技术的引领者,近年来SMART(Switching Mechanism at 5’ End of RNATemplate)技术被公认为是微量RNA和单细胞RNA-seq的行业金标准。现在创新的DNASMART技术将模板转换机制的优势拓展到了DNA分析,利用DNA SMART ChIP-Seq Kit无需接头连接即可构建ChIP测序文库,极大地缩短了实验进程。
但是科学技术的发展有时需要突破实验技术的挑战,那么如何从染色质免疫共沉淀(ChIP)实验中获得的少量DNA制备新一代测序(NGS)文库就是科研人员面临的挑战之一。
大部分ChIP测序文库的制备需要测序接头连接,但是这些制备方法的起始材料仅限于双链DNA。DNASMART ChIP-Seq Kit提供了一种强大、可靠的DNA ChIP-seq方法,尤其适用于低起始量材料(100 pg–10ng),整个文库制备过程只需4小时,不仅适用于dsDNA也适用于ssDNA,是DNA CHIP-Seq的理想选择。
DNA SMART技术
DNA SMART技术传承了SMART技术的优势,无需接头连接和纯化,整个过程依赖于SMARTScribe ReverseTranscriptase(RT),该RT可以复制DNA模板并在新合成的DNA 3’端添加几个核苷酸,巧妙设计的DNA SMARTOligonucleotide的末端可与这几个额外的核苷酸互补延伸DNA模板,SMARTScribe Reverse Transcriptase可以继续复制模板至oligonucleotide的末端从而在新合成的DNA末端直接添接头序列。利用含有Illumina测序接头的引物通过PCR扩增即可获得测序文库。
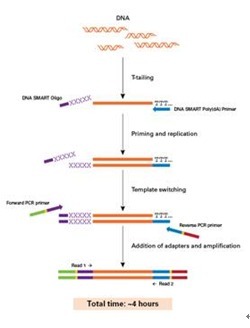
优化的实验流程
DNA SMART ChIP-Seq Kit采用了PCR扩增制备文库后进行片段长度选择和纯化的一体化流程,与其他采用PCR扩增前进行片段长度选择的方法相比,PCR后片段筛选可以在获得高质量文库的同时获得高产量文库。
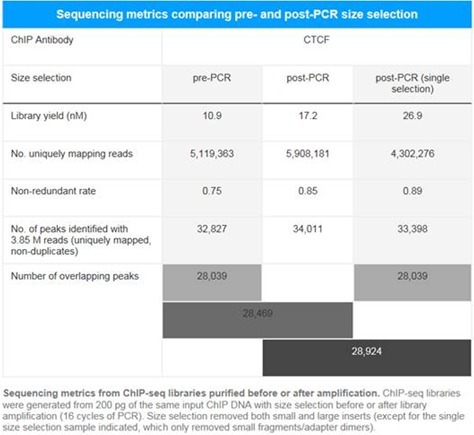
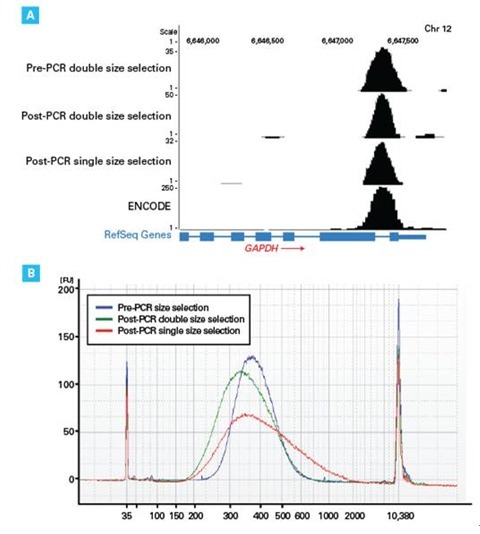
扩增后进行片段长度选择并不会影响测序数据的质量,Peaks的分布和形态与报道的ENCODE数据吻合。
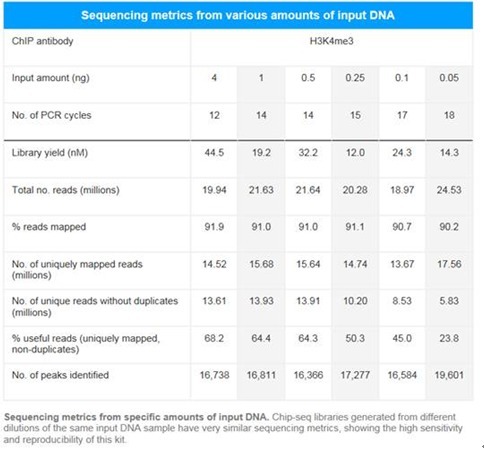
高灵敏度文库制备
DNA SMART ChIP-Seq Kit可以极微量的片段化DNA为起始制备测序文库。以不同起始量材料制备的文库都可以获得很高数量的特异的、非重复读段,并且不同起始量文库均可获得相近的峰数。
利用DNA SMART ChIP-Seq Kit可以制备重复性很高的测序文库,起始量100 pg以上时,相同起始量制备的技术重复文库可以获得>93%的重叠,不同起始量制备的文库之间的重叠>94%。

ChIP-seq library complexity and reproducibility is maintained acrossinput amounts. The reproducibility between technical replicates wassimilar across input amounts (Panel A). The non-redundant rate(normalized for 10 million uniquely mapped reads) was well above thestandard recommended by the ENCODE project (0.8) for inputs >0.5 ng(Panel B; error bars indicate the standard deviation of two technicalreplicates). Compared to the 4 ng library, the number of peaks weresimilar across lower input libraries (Panel C). The shape and locationof the peaks was similar across input levels, and matched very well toENCODE data (293 cells, anti-H3K4me3 antibody, U. Washington), even foras little as 50 pg input DNA (Panel D).
DNA SMART ChIP-Seq Kit基于改良的模板转移机制,无需接头连接即可制备测序文库,兼容ssDNA和dsDNA,是微量DNAChIP-seq的理想选择。